利用oligo 7.57设计引物(等位基因特异性PCR)
1、打开oligo 7.57,分别单击File——New Sequence,在打开的窗口中输入DNA序列,然后单击序列上面的勾就OK了。
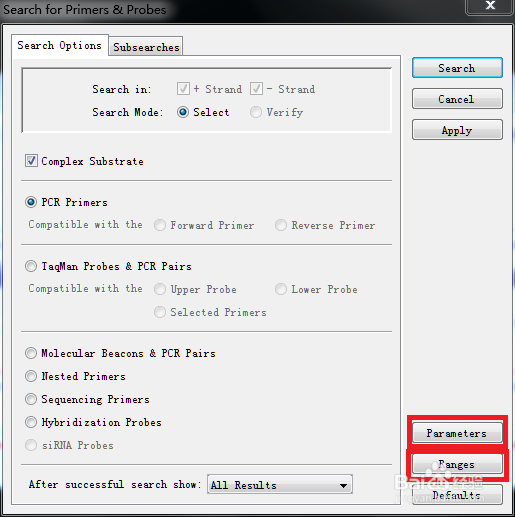
2、单击菜单栏的Search——for Primers & Probes...
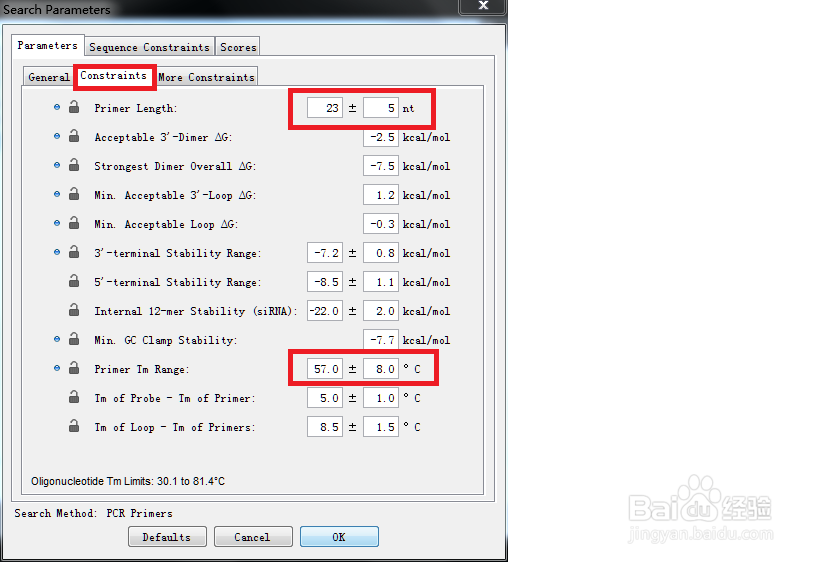
3、分别单击 Parameters 和 Range衡痕贤伎s 设计引物的参数和范围。接下来先单击Parameters。只需修改引物长度和Tm值的范围即可,如下方第一张图。另外因为突变位点是确定的,所以要确定下游引物的位置,如下方第二张图所示,即固定了3'端的位置(这里笔者是将突变位点处的引物作为下游引物)
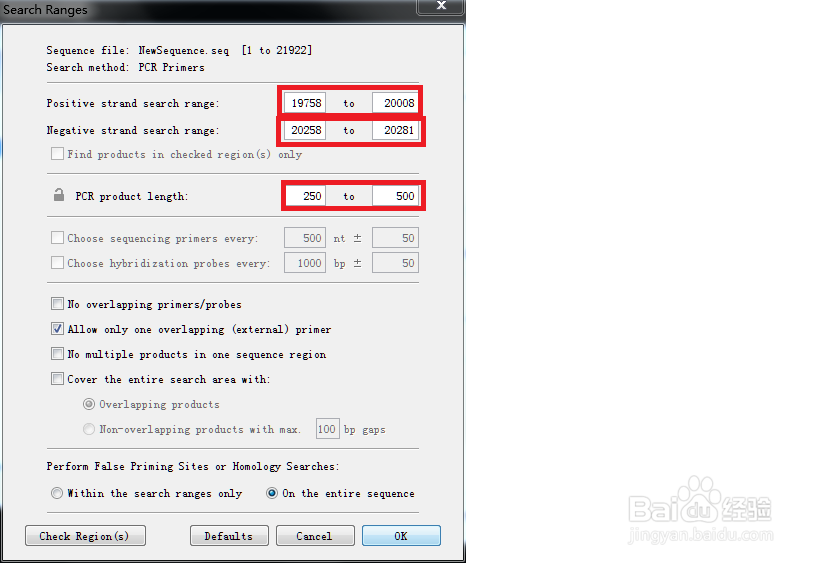
4、再单击Ranges,设计范围如下图。一般PCR产物长度250——500bp为宜。注:该突变位点在20258bp的位置。
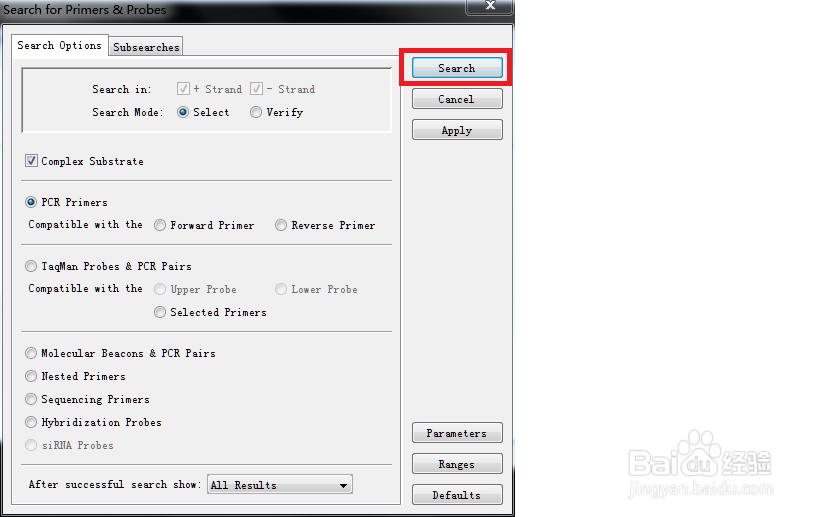
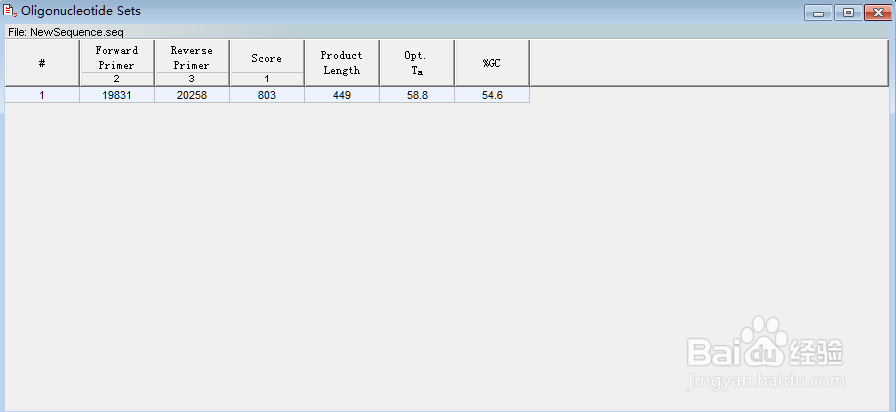
5、单击Search,如下方第一张图。跳出搜索结果,如下方第二张图。
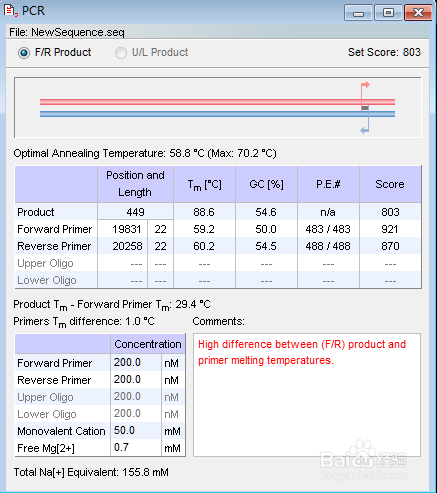
6、双击上方第二张图中搜索到的引物,跳出的窗口如下图,是该对引物的基本信息,包括上下游的位置,长度,Tm值,GC含量,分数等。
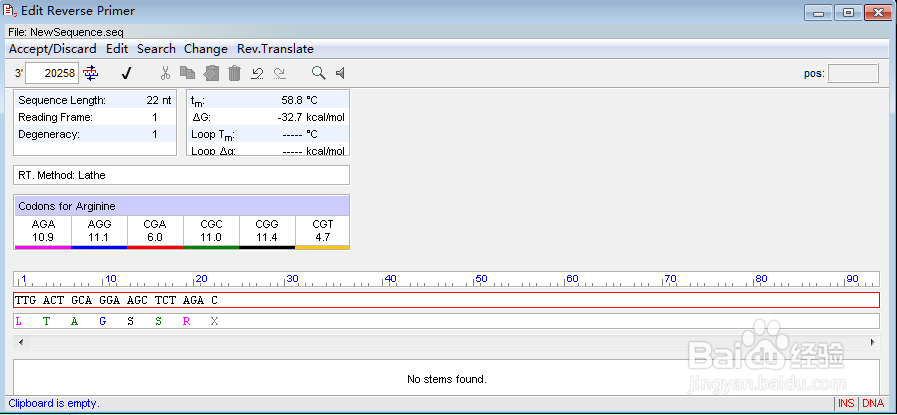
7、单击菜单栏Edit——Reverse Primer,打开下方第一张图的界面,将3’端第三或第四位的碱基改成错配(这里是将第四位的碱基G改为A,因为这样改可以取消下游引物的发夹结构),改好后如下方第二张图。并单击第二张图菜单栏下方的勾。
8、至此引物但诌诎箬已经设计完了,接下来就可以在菜单栏Analyze下对该对引物进行分析,主要分析该对引物的发夹结籁缰汾秘构,二聚体,错配情况。当然也应该注意一下上下游引物的Tm值,GC含量等是否合适,如果不合适的话,可以修改引物相关参数再进行设计或修改错配碱基来找到合适的引物对。至于引物分析我这里就不做详细介绍了。当你设计好第一对引物后,就应该设计另一条下游引物了,只需将突变位点的碱基改成相应的碱基就行了(这里的碱基修改是根据你该突变位点是什么碱基突变成什么碱基来决定的),上游引物不变。同理再进行引物分析。